Chapter 12 Maskinslæring i tidyverse






library(rsample) #install this
library(tidyverse)
library(ranger) #install this, random forest
library(palmerpenguins)“Absorber hvad der er nyttigt, afvis det, der er ubrugeligt, tilføj det, der specifikt er dit eget.” - Bruce Lee
12.1 Indledning og læringsmålene
Her bliver en lynhurtig introduktion til maskinslæring koncepter - emnet er i virkelighed kæmpe stort og jeg springer over mange detaljer her. Men jeg håber, at det giver en god grundlag/forståelse af den overordnet ramme, og at det kan være med til at øge forståelsen af de mange artikler i biologiske område der benytter maskinslæring modeller, samt motivere dig til at eventuelle tage yderligere kurser i området.
I emnet i dag introducerer jeg den grundlæggende ramme hvor man udvikler en model på træning data med cross-fold-validation og dernæst vurderer den model på testing data, som ikke var en del af selve træningsprocessen. Jeg inddrager også en classification model, som vi både optimerer (“tune”) og bruger til at forudsige hvem, der overlevede tradgedien på titanic!
Jeg har bestræbt på at arbejde så vidt som muligt kun indenfor for den tidyverse ramme og sæt funktioner vi allerede har dækket i kurset, som gør det nemmere at forstå mæskingslæring processen uden at blive distraheret af mange nye pakker og funktioner. Processen virker måske lidt indviklet i starten, men generaliseres meget og nemt kan udvides i mange kontekster og situationer - du kan nærmest tage eget data og bare tilpasse min kode fra enten den regression eller classification scenarie nedenunder.
12.1.1 Læringsmålene
Du skal være i stand til at
- Kende anvendelse af testing og training datasæt og beskrive cross-fold-validation processen
- Anvende 5-fold-cross validation til at udvikle en model og beregner statistikker til at evaluere modellen
- Lave en classification model for at forudsige hvem der overlevede titanic
- Lave “tuning” af en parameter og bestemme om en “final” model
12.2 Video ressourcer
- Video 1: Data splitting and cross-fold validation i en regression kontekst
- Video 2: Cross-fold validation i en classifcation contekst (random forest)
- Video 3: Tuning af parametre i classification og variabel importance
12.3 Regression models
Vi starter med at bygge processen op i en kontekst som vi godt kender i forvejen - en lineær regression model. Til det formål benytter vi endnu engang datasættet penguins, og målet er: hvor godt kan man forudsige variablen body_mass_g i de forskellige pingviner?
library(palmerpenguins)
penguins <- penguins %>% drop_na()12.3.1 Oprette testing and training sets
Hvordan kan man vide, om en model er god til at forudsige hvad der sker når der kommer nye observationer? Det vil sige, fungerer modellen godt kun i forhold til observationerne som blev brugt til at opbygge selve modellen (så kaldt “overfitting”), eller kan modellen generalises til andre, ligenende situationer? For at vide det kan man overvejer at splitte datasættet før man lave modellen - det vil sige et datasæt som man bruger til at opbygge modellen, og et datasæt som man kan bruge som “nye observationer”, hvor man kan teste, hvor godt modellen egentlig er (fordi i datasættet kender vi faktisk de virkelige værdier som vi kan sammenligne med vores forudsigelser fra modellen).

Pakken rsample fra tidymodels har funktionen initial_split, der opdeler datasættet således at fk. 75% af observationerne hører til det “training” sæt og 25% hører til det “testing” sæt:
penguins_split <- initial_split(penguins, prop = 0.75)
penguins_split## <Analysis/Assess/Total>
## <249/84/333>Det betyder, at ud af de 333 observationerne, bliver 249 brugt som “training” og 84 er beholdt tilbage som en “testing” sæt. Jeg udtrækker bagefter de to sæt til at få “penguins_training” og “penguins_test” som i følgende:
penguins_training <- penguins_split %>% training()
penguins_test <- penguins_split %>% testing()Jeg vil gerne anvende penguins_training til at opbygge modellen og så kun kigge på penguins_test senere for at evaluere modellens evne til at lave forudsigelser.
12.3.2 Cross-fold validation
For at udvikle min model med mit træning datasæt anvender jeg “5-cross-fold-validation”. I 5-fold-cross-validation splitte man et datasæt op i 5 stykker (“folds”). Man træner dernæst en model på 4 af de 5 stykker (folds), og tester på den sidste stykke (der ofte kaldes for “validation” sæt, og som ikke selv var en del af datasættet brugte til at fitte selve modellen). Man gentager model “training” processen i alt fem gange, hvor hvert styk er brugt præcis én gang som “valiation” sæt.
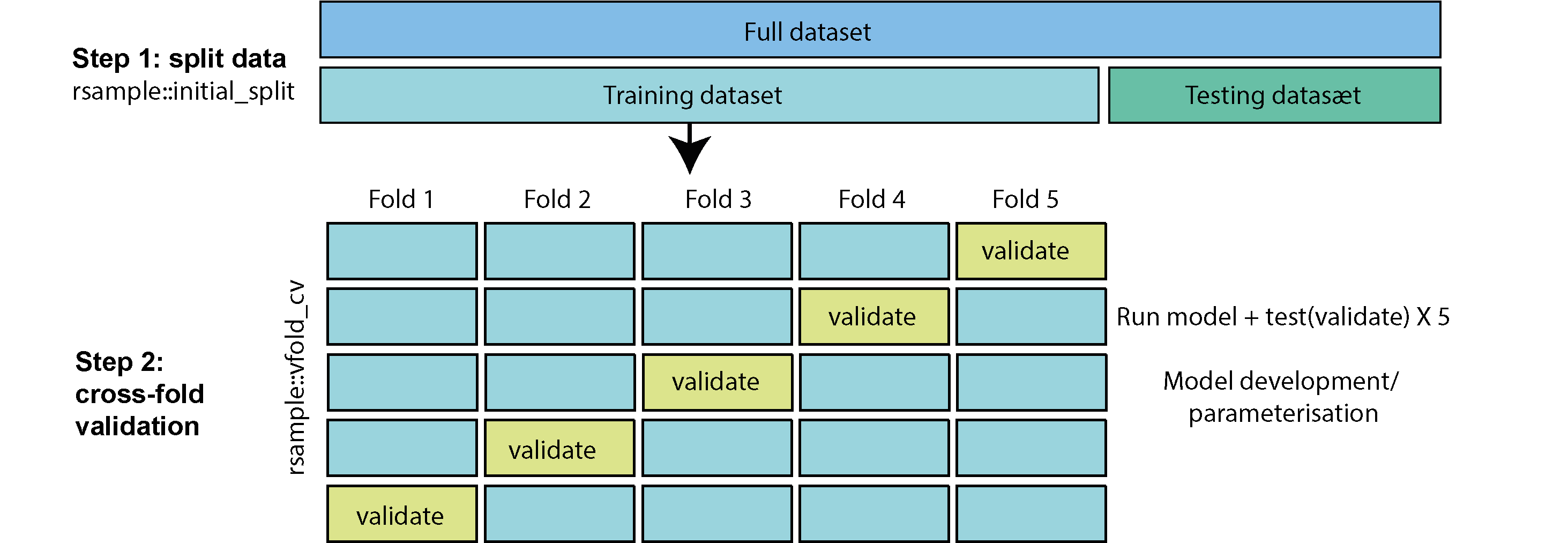
Tilgangen har mange fordele. Hvis der var for eksempel kun én testing sæt og ved tilfældighed har den sæt observationer som er især nemme at forudsige, så får vi en fejlagtig fornemmelse at modellen er bedre end den er i virkeligheden - vi undgår det vi at bruge 5 forskellige validation sæt som dækker hele træning sæt. Vi kan således lede efter en kommination af parametre, der konsekvent giver en god model over samtlig validation sæt - vi kan være mere sikker på at få en model, der virker godt i vores testing sæt til sidste.
Der er en nem funktion man kan bruge i rsample-pakken, der hedder vfold_cv til at lave cross validation. Her angiver jeg v=5 for at vise, at jeg gerne vil have 5-folds.
cv_split <- vfold_cv(penguins_training, v = 5)
cv_split## # 5-fold cross-validation
## # A tibble: 5 × 2
## splits id
## <list> <chr>
## 1 <split [199/50]> Fold1
## 2 <split [199/50]> Fold2
## 3 <split [199/50]> Fold3
## 4 <split [199/50]> Fold4
## 5 <split [200/49]> Fold5Så kan man se, at vi får en dataframe med 5 datasæt som er lagret i kolonnen splits. Jeg vil gerne udtrække de forskellige “training” og “testing”(/“validate”) sæt ud i deres egne kolonner, som i følgende med map indenfor mutate:
cv_data <- cv_split %>%
mutate(
train = map(splits, ~.x %>% training), #extract training datasets
validate = map(splits, ~.x %>% testing) #extract validation datasets
)
cv_data## # 5-fold cross-validation
## # A tibble: 5 × 4
## splits id train validate
## <list> <chr> <list> <list>
## 1 <split [199/50]> Fold1 <tibble [199 × 8]> <tibble [50 × 8]>
## 2 <split [199/50]> Fold2 <tibble [199 × 8]> <tibble [50 × 8]>
## 3 <split [199/50]> Fold3 <tibble [199 × 8]> <tibble [50 × 8]>
## 4 <split [199/50]> Fold4 <tibble [199 × 8]> <tibble [50 × 8]>
## 5 <split [200/49]> Fold5 <tibble [200 × 8]> <tibble [49 × 8]>12.3.3 Bygge modellen og lave forudsigelser
Vi vil gerne lave en model på alle vores datasæt i kolonnen train hver for sig, og forudgsige body_mass_g i hvert tilknyttet validation sæt. Vi kan gøre det med en custom funktion som i følgende:
my_lm_func <- ~lm(body_mass_g ~ ., data = .x) # . betyder alle variabler i .x er uafhængige untaget body_mass_g
cv_models_lm <- cv_data %>%
mutate(model = map(train, my_lm_func))
cv_models_lm## # 5-fold cross-validation
## # A tibble: 5 × 5
## splits id train validate model
## <list> <chr> <list> <list> <list>
## 1 <split [199/50]> Fold1 <tibble [199 × 8]> <tibble [50 × 8]> <lm>
## 2 <split [199/50]> Fold2 <tibble [199 × 8]> <tibble [50 × 8]> <lm>
## 3 <split [199/50]> Fold3 <tibble [199 × 8]> <tibble [50 × 8]> <lm>
## 4 <split [199/50]> Fold4 <tibble [199 × 8]> <tibble [50 × 8]> <lm>
## 5 <split [200/49]> Fold5 <tibble [200 × 8]> <tibble [49 × 8]> <lm>Nu vil vi gerne bruge datasættet i kolonnen validate til at lave en forudsigelse til hver af vores modeller. Til det formål kan man bruge funktionen predict() - man tager en model samt et nye datasæt (dvs. tilsvarende datasæt i kolonne validate), og så få nogle forventede body_mass_g værdier ud fra modellen anvendte på de nye data. I følgende bliver disse forventede “predicted” værdier til body_mass_g lagret i kolonnen validate_predicted. Da vi faktisk vide de virkelige værdier til body_mass_g i vores validation datasæt laver jeg også en kolonne validate_actual som er disse virkelige værdier, som jeg kan sammenligne med mine forventede værdier.
cv_prep_lm <- cv_models_lm %>%
mutate(
actual_mass = map(validate, ~.x %>% pull(body_mass_g)), #actual body_mass_g values for validation set
predicted_mass = map2(.x = model, .y = validate, ~predict(.x, .y)) #predicted body_mass_g values for validation set
)12.3.4 Vurdere resultater på validation sæt
Jeg vil gerne sammenligne mine nye forudsigelser direkte med mine virkelige værdier til body_mass_g, hvilket kan give os en indikation på, hvor godt vores modeller egentlig er. I følgende tager jeg id, actual_mass og predict_mass ud fra dataframen cv_pred_lm og gennem dem i en ny dataframe prediction_df:
prediction_df <- cv_prep_lm %>% select(id,actual_mass,predicted_mass) %>% unnest(c(actual_mass,predicted_mass))Til hver af de fem folds bregener jeg statisttiken “mean absolute error” der tager afstanden mellem de forventede værdier (predicted_mass) og virkelige værdier (actual_mass) og tager dernæst middelværdien (over deres absolute værdier dvs. uden minus tegn). Jo mindre “mae” er, jo bedre modellen ser ud.
#smaller mae = better model
mae_df <- prediction_df %>% group_by(id) %>% summarise("mae" = mean(abs(actual_mass-predicted_mass)))Her er et plot af statistikken “mae” til de forskellige folds. I gennemsnit er det 233.0974471, der betyder at vores forudsigelser er ude/forkert ved knap 300 g i gennemsnit - ikke dårligt når den gennemsnits pingvin vejer 4207.06 g.
mae_df %>%
ggplot(aes(y=mae,x=id,fill=id)) +
geom_bar(stat="identity",colour="black") +
theme_bw()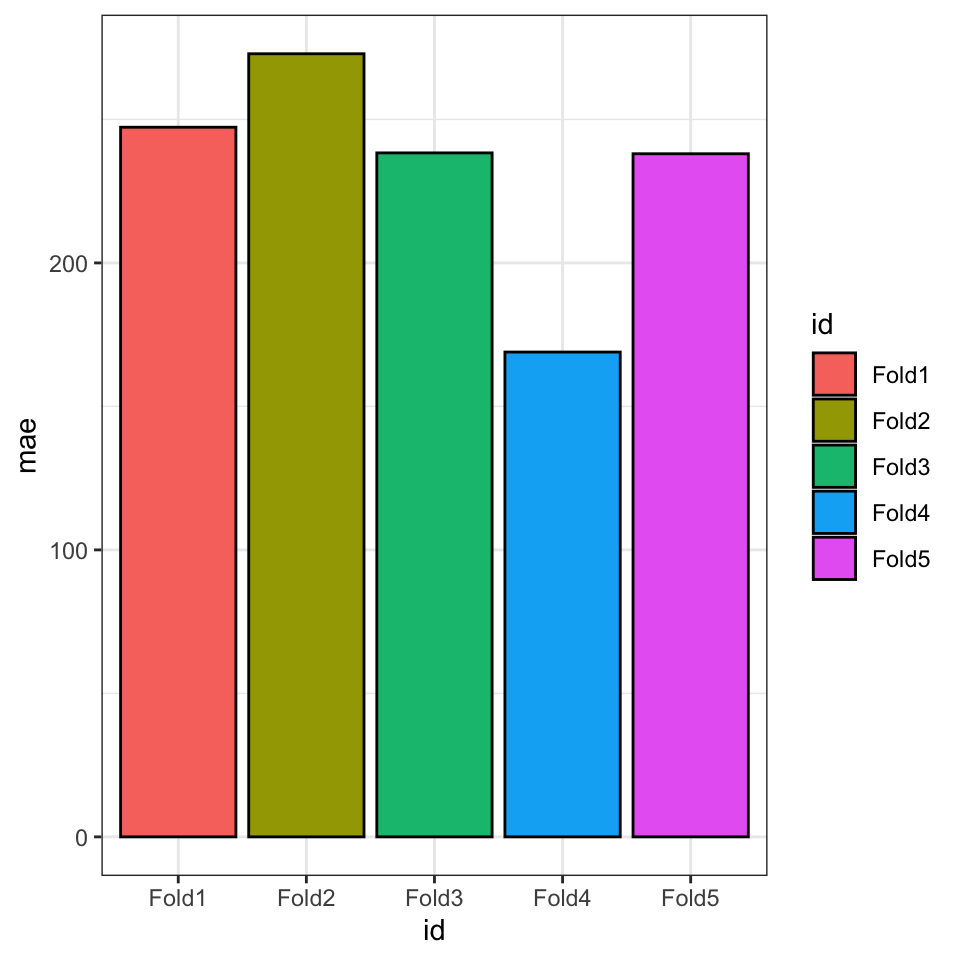
12.3.5 Teste og evaluere “final” model
Vi har lavet forudsigelser over de forskellige stykker i vores træning sæt, men vi har ikke endnu lavet forudsigelser på vores testing sæt penguins_test, som blev holdt ud af hele træningsprocessen. Efter den cross-validation proces skal man lave en “final” model, der bruger samtlige training data på én gang (dvs. ikke bare en enkel fold, der vi gerne vil opbygge den endelige model fra så meget data, som muligt. Bemærk at når det er kun et datasæt, behøver vi ikke at bruge en custom funktion.
model_final <- lm(body_mass_g ~ ., data = penguins_training)Nu kan vi endelige anvende vores testing data (som vi holdt ud før cross-validaiton processen): her anvender jeg min “final model” til at forudsige body_mass_g på pingvinerne i datasættet penguins_test. Dernæst sammenligner vi vores forudsigelser med den virkelige værdier ligesom i ovenstående (men her har jeg ikke forskellige folds at tage i betragtning).
prediction_df <- tibble("actual_mass"=penguins_test %>% pull(body_mass_g),
"predicted_mass"=predict(model_final,penguins_test))
prediction_df %>% summarise("mae" = mean(abs(actual_mass-predicted_mass)))## # A tibble: 1 × 1
## mae
## <dbl>
## 1 235.Man kan se, at vores final model lave forudsigelser som er i gennemsnit 235.5g væk fra pingvinernes virkelige vægt (det vil sige, at forudsigelsen kan være over eller under den virkelige værdi, men i gennemsnit er afstanden fra den virkelige værdi 230g).
12.4 Classification models
I ovenstående proces lavede vi en regression model, hvor afhængig variabel er kontinuerly (body_mass_g). I en classification model er den afhængig variabel kategorisk (fk. “yes” eller “no” i forhold til variblen Survived i datasættet titanic). Vi viser her at det er meget nemt at skifte fra den ene type model til en anden og bruger stort set samme kode (kun små ændringer).
Ovenstående ramme også giver os muligheden for at opbygge modeller, hvor der faktisk er forskellige parametre, der skal vælges. I ovenstående eksempel med penguins var der faktisk ikke meget “tuning” vi skulle lave for at udvikle modellen. Jeg viser her hvordan man kan “tune” en parameter indenfor samme ramme i vores classification model.
Jeg åbner først titanic datasæt. Datasættet i titanic-pakken er allerede blevet adskilt efter training og testing men lad os bare ignorerer det og følger samme ramme som i ovenstående.
library(titanic)
titanic <- titanic_train
titanic <- titanic %>% drop_na() %>% as_tibble() %>%
mutate(Survived = factor(if_else(Survived == 1, "yes", "no"),levels=c("yes","no"))) %>%
mutate(Pclass = recode(Pclass, `1`="first",`2`="second",`3`="third")) %>%
mutate(Pclass = as_factor(Pclass),
Sex = as_factor(Sex),
Port = as_factor(Embarked),
Solo = if_else(SibSp + Parch == 0, "yes", "no")) %>%
select(Survived, Pclass, Sex, Age, Solo, Port, Fare)
titanic## # A tibble: 714 × 7
## Survived Pclass Sex Age Solo Port Fare
## <fct> <fct> <fct> <dbl> <chr> <fct> <dbl>
## 1 no third male 22 no S 7.25
## 2 yes first female 38 no C 71.3
## 3 yes third female 26 yes S 7.92
## 4 yes first female 35 no S 53.1
## 5 no third male 35 yes S 8.05
## 6 no first male 54 yes S 51.9
## 7 no third male 2 no S 21.1
## 8 yes third female 27 no S 11.1
## 9 yes second female 14 no C 30.1
## 10 yes third female 4 no S 16.7
## # … with 704 more rowsHer vil vi gerne forudsige hvem der overlevede, og hvem der ikke overlevede. Det er et eksempel på et “classification” problem, hvor der er to mulige svar (“yes” eller “no”) og man gerne vil beregne sandsynlighed for de to muligheder. Det er hvad der kaldes for et “supervisered” problem, fordi når vi træner modellen, ved vi faktisk hvem der overlevede i datasættet. Det er til forskel for eksempelvis kmeans clustering, hvor vi ikke har svaret i forvejen (men bare kan gætte, at de forskellige clusters relatere til noget, for eksempel hvilke species en observation hører til).
Før vi går i gang, her er et overblik over processen med at opbygge modellen med titanic:
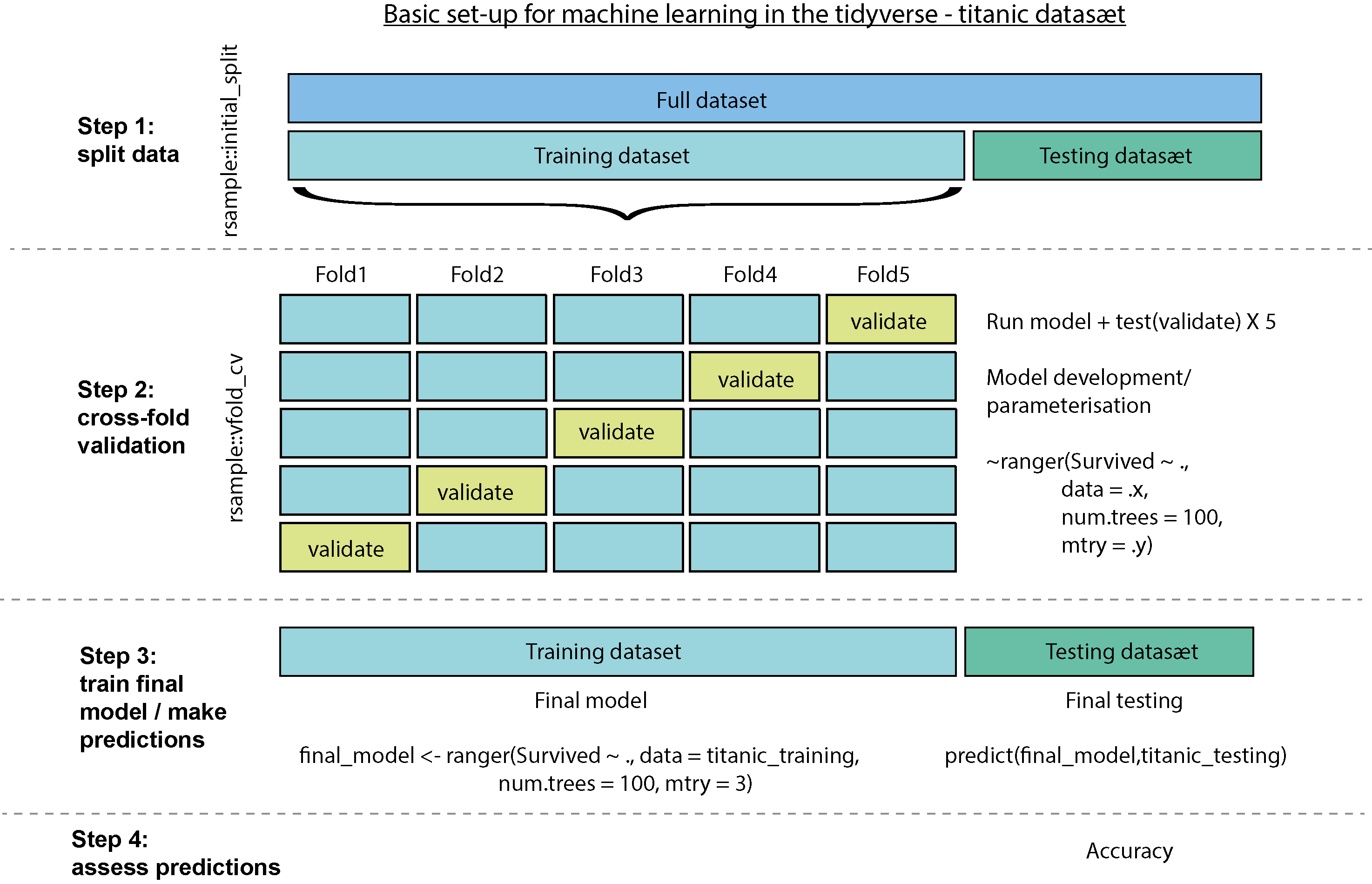
I følgende steps forklarer jeg processen med titanic i flere detaljer:
12.4.1 Splitte datasættet og lave cross-validation dataframe
set.seed(333)
titanic_split <- initial_split(titanic, prop = 0.75)
titanic_training <- titanic_split %>% training()
titanic_test <- titanic_split %>% testing()Vi anvender 5-fold cross validation på datasættet titanic_training og udtrækker de træning og testing sæt til de forskellige folds (og lagrer dem i henholdvis train og validate i dataframen cv_data):
set.seed(333)
cv_split <- vfold_cv(titanic_training, v = 5)
cv_data <- cv_split %>%
mutate(
train = map(splits, ~.x %>% training()),
validate = map(splits, ~.x %>% testing())
)
cv_data## # 5-fold cross-validation
## # A tibble: 5 × 4
## splits id train validate
## <list> <chr> <list> <list>
## 1 <split [428/107]> Fold1 <tibble [428 × 7]> <tibble [107 × 7]>
## 2 <split [428/107]> Fold2 <tibble [428 × 7]> <tibble [107 × 7]>
## 3 <split [428/107]> Fold3 <tibble [428 × 7]> <tibble [107 × 7]>
## 4 <split [428/107]> Fold4 <tibble [428 × 7]> <tibble [107 × 7]>
## 5 <split [428/107]> Fold5 <tibble [428 × 7]> <tibble [107 × 7]>12.4.2 Definere classification model
Næste vil vi gerne anvende en model over de fem training sæt. Et godt eksempel på en classifikation metode som er både nem at bruge og meget kræftig er en “random forest”. Her er det ikke nødvendeigt at forstå al detaljer, men helt kort - man opbygger beslutningstræer baserede på tilfældigt udvalgte subsets af data. I et beslutningstræ laver man “splits” over de forskellige variabler, som man bruger for at bestemme, om en passagerer overlevede eller ej (se billede). Når man kan gøre samme process flere gange, over eksempelvis 100 træer, der involverer 100 tilfældige subsæt af data, kan man tage et gennemsnit, som udgør de endelige beslutninger (predictions eller forudsigelser).
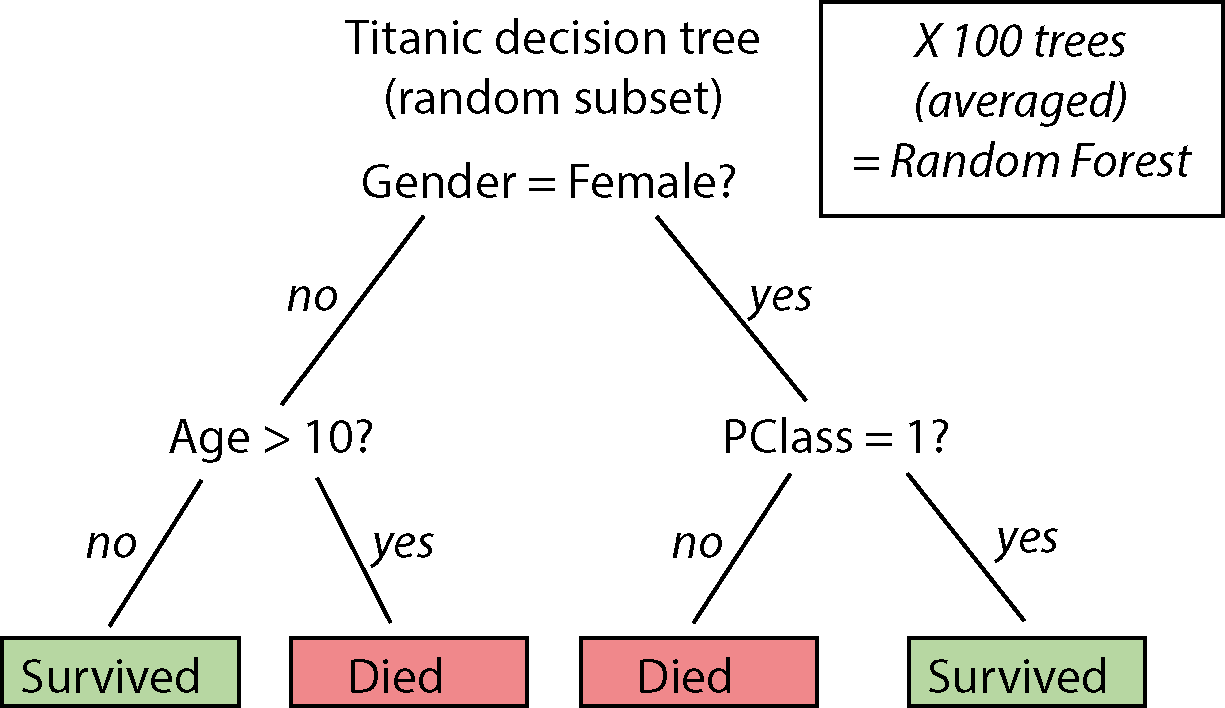
I følgende anvender jeg funktionen ranger() (installer pakken ranger), der fitter en random forest i R på datasætet.
set.seed(333)
ranger_model <- ~ranger(formula = Survived ~ . , # . betyder all variabler i datasættet som uafhængig
data = .x,
num.trees = 100)Lidt om ranger-modellens parametre:
- Jeg angiver en formel - variablen
Surviveder afhængig og alle andre variabler er uafhængig (betegnet ved “.” for at spare tid). - data er
.xog referer til en træning sæt til én af vores “folds” i vores cross-fold validation datasæt. num.treesfortæller, hvor mange træer jeg vil lave (jo mere jo bedre, men der er en trade-off med hensyn til tid/energi)
12.4.3 Anvende modellen og lave predictions
Jeg laver mine modeller ved at oprette en ny kolonne model i dataframe cv_data, hvor jeg anvender min custom funktion med map:
cv_models_rf <- cv_data %>%
mutate(model = map(train, ranger_model))
cv_models_rf## # 5-fold cross-validation
## # A tibble: 5 × 5
## splits id train validate model
## <list> <chr> <list> <list> <list>
## 1 <split [428/107]> Fold1 <tibble [428 × 7]> <tibble [107 × 7]> <ranger>
## 2 <split [428/107]> Fold2 <tibble [428 × 7]> <tibble [107 × 7]> <ranger>
## 3 <split [428/107]> Fold3 <tibble [428 × 7]> <tibble [107 × 7]> <ranger>
## 4 <split [428/107]> Fold4 <tibble [428 × 7]> <tibble [107 × 7]> <ranger>
## 5 <split [428/107]> Fold5 <tibble [428 × 7]> <tibble [107 × 7]> <ranger>Næste laver jeg forudsigelser over de fem validate datasæt med funktionen predict() - det fungere på samme måde som i ovenstående regression model, men med ranger er man nødt til at udtrække selve forudsigelserne ved at tilføje $predictions, ligesom i følgende funktion my_prediction_function:
my_prediction_function <- ~predict(.x, .y)$predictions
cv_prep_rf <- cv_models_rf %>%
mutate(
actual = map(validate, ~.x %>% pull(Survived)), #virkelig "yes" eller "no"
predicted = map2(.x = model, .y = validate,my_prediction_function) #forudsigelser af "yes" eller "no"
)Nu lad os kigge på vores forudsigelser (“yes” eller “no”) ved siden af de virkelige værdier (“yes” eller “no”) i variablen Survived, for hver af de fem validation sæt:
predictions_df <- cv_prep_rf %>% select(id, actual,predicted) %>% unnest(c(actual,predicted))
predictions_df## # A tibble: 535 × 3
## id actual predicted
## <chr> <fct> <fct>
## 1 Fold1 yes yes
## 2 Fold1 yes yes
## 3 Fold1 no no
## 4 Fold1 yes yes
## 5 Fold1 no no
## 6 Fold1 yes yes
## 7 Fold1 yes no
## 8 Fold1 no no
## 9 Fold1 yes yes
## 10 Fold1 yes yes
## # … with 525 more rows12.4.4 Beregne accuracy
Hvor mange fik vi rigtige? Vi beregner “accuracy” som måler proportionen af “rigtig” forudsigelser (både actual og predicted er enige om, hvem der overlevede eller ikke overlevede). Da jeg har forudsigelser til 5 forskellige datasæt (validation sæt for hver “fold”), anvender jeg group_by(id), og dernæst summary til at beregne proportionen af tilfælde variablen actual er den samme som predicted.
my_accuracies <- predictions_df %>%
group_by(id) %>%
summarise("accuracy" = sum(actual==predicted)/n())
my_accuracies## # A tibble: 5 × 2
## id accuracy
## <chr> <dbl>
## 1 Fold1 0.738
## 2 Fold2 0.813
## 3 Fold3 0.841
## 4 Fold4 0.822
## 5 Fold5 0.813Jeg kan omsætte tallene til et plot:
my_accuracies %>%
ggplot(aes(x=id,y=accuracy,fill=id)) +
geom_bar(stat="identity",colour="black") +
theme_bw()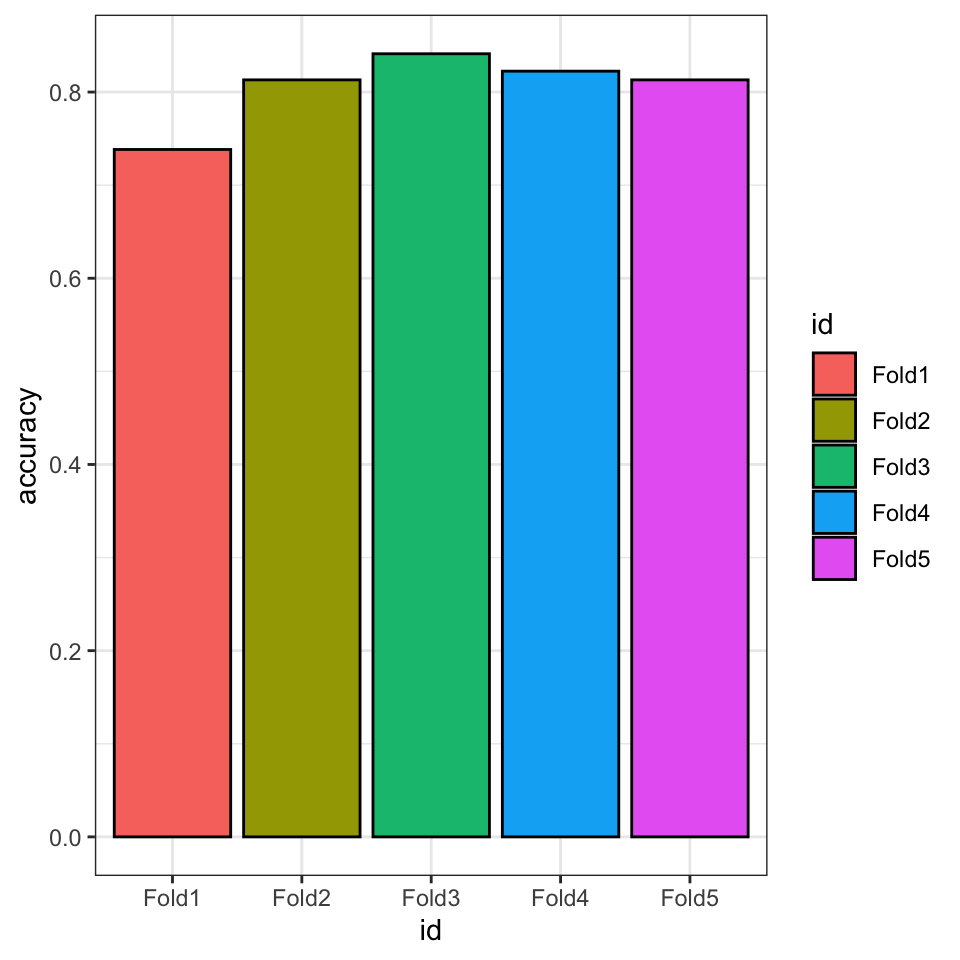
12.4.5 Parameter tuning
I funktionen ranger var der faktisk nogle parametre som kan have indfyldelse over modellens predictions til sidste - én af dem er for eksempel num.trees - hvis man bruger fk. 100 træer får man sandsynligvis bedre resultater end hvis man anvender kun et træ. En anden parameter, som vi angav som “default” i ovenståene analyse er mtry. Det fortæller hvor mange variabler vi skal inddrage i en enkel beregning i træerne (det inddrager mere tilfældighed i altgoritmen og undgår, at 1-2 variabler har for meget indfyldelse på resulaterne).
Vi vil gerne prøve de forskellige værdier til mtry i modellen og se, hvilken værdi giver bedste resultater. I tidyr-pakken er der en nyttig funktion crossing, der laver kopier af de forskellige folds datasæt, én til hver mulig værdie af mtry:
# Prepare for tuning your cross validation folds by varying mtry
cv_tune <- cv_data %>%
crossing(mtry = c(2:6))
cv_tune## # A tibble: 25 × 5
## splits id train validate mtry
## <list> <chr> <list> <list> <int>
## 1 <split [428/107]> Fold1 <tibble [428 × 7]> <tibble [107 × 7]> 2
## 2 <split [428/107]> Fold1 <tibble [428 × 7]> <tibble [107 × 7]> 3
## 3 <split [428/107]> Fold1 <tibble [428 × 7]> <tibble [107 × 7]> 4
## 4 <split [428/107]> Fold1 <tibble [428 × 7]> <tibble [107 × 7]> 5
## 5 <split [428/107]> Fold1 <tibble [428 × 7]> <tibble [107 × 7]> 6
## 6 <split [428/107]> Fold2 <tibble [428 × 7]> <tibble [107 × 7]> 2
## 7 <split [428/107]> Fold2 <tibble [428 × 7]> <tibble [107 × 7]> 3
## 8 <split [428/107]> Fold2 <tibble [428 × 7]> <tibble [107 × 7]> 4
## 9 <split [428/107]> Fold2 <tibble [428 × 7]> <tibble [107 × 7]> 5
## 10 <split [428/107]> Fold2 <tibble [428 × 7]> <tibble [107 × 7]> 6
## # … with 15 more rowsNu kan jeg bygge en model op til hver kombination af fold (variablen id) og mtry (variablen mtry). Bemærk at jeg bruger map2 her, der jeg gerne vil bruge to kolonner af min næsted dataframe - mtry (betegnet som .y i funktionen) og train (betegnet som .x i funktionen).
# Build a model for each fold & mtry combination
my_ranger_func <- ~ranger(formula = Survived~., data = .x,
mtry = .y,
num.trees = 100)
cv_models_rf <- cv_tune %>%
mutate(model = map2(train, mtry, my_ranger_func))Og utrækker predictions med predict ligesom i ovenstående:
cv_prep_rf <- cv_models_rf %>%
mutate(
actual = map(validate, ~.x$Survived),
predicted = map2(.x = model, .y = validate, ~predict(.x, .y)$predictions)
)Nu laver jeg en dataframe ligesom i ovenstående som viser mine predictions - men denne gange har jeg predictions over både min folds og de forskellige værdier for “mtry”:
predictions_df <- cv_prep_rf %>% select(id,mtry,actual,predicted) %>% unnest(cols=c(actual,predicted))
predictions_df## # A tibble: 2,675 × 4
## id mtry actual predicted
## <chr> <int> <fct> <fct>
## 1 Fold1 2 yes yes
## 2 Fold1 2 yes yes
## 3 Fold1 2 no no
## 4 Fold1 2 yes yes
## 5 Fold1 2 no no
## 6 Fold1 2 yes yes
## 7 Fold1 2 yes no
## 8 Fold1 2 no no
## 9 Fold1 2 yes yes
## 10 Fold1 2 yes yes
## # … with 2,665 more rowsJeg anvender group_by efter både mtry og id for at beregne accuracy:
my_accuracies <- predictions_df %>%
group_by(mtry,id) %>%
summarise("accuracy" = sum(actual==predicted)/n()) %>%
ungroup(mtry)## `summarise()` has grouped output by 'mtry'. You can override using the
## `.groups` argument.I følgende plot visualiseres den sammenlagt accuracy over all folds, til hver mulig værdi af “mtry”-parameteren. Man kan se, at det gør ikke meget forskel, hvilken værdie af “mtry” jeg vælger i min final moodel, min den højeste sammenlagt søjle hører til “mtry=3” - så jeg tager 3 som min “mtry” parameter i min “final” model.
my_accuracies %>% ggplot(aes(fill=id,x=mtry,y=accuracy)) +
geom_bar(stat="identity",colour="black") +
ylab("total accuracy over all folds") + ggtitle("Total accuracy stacked up over the 5 folds") +
theme_classic()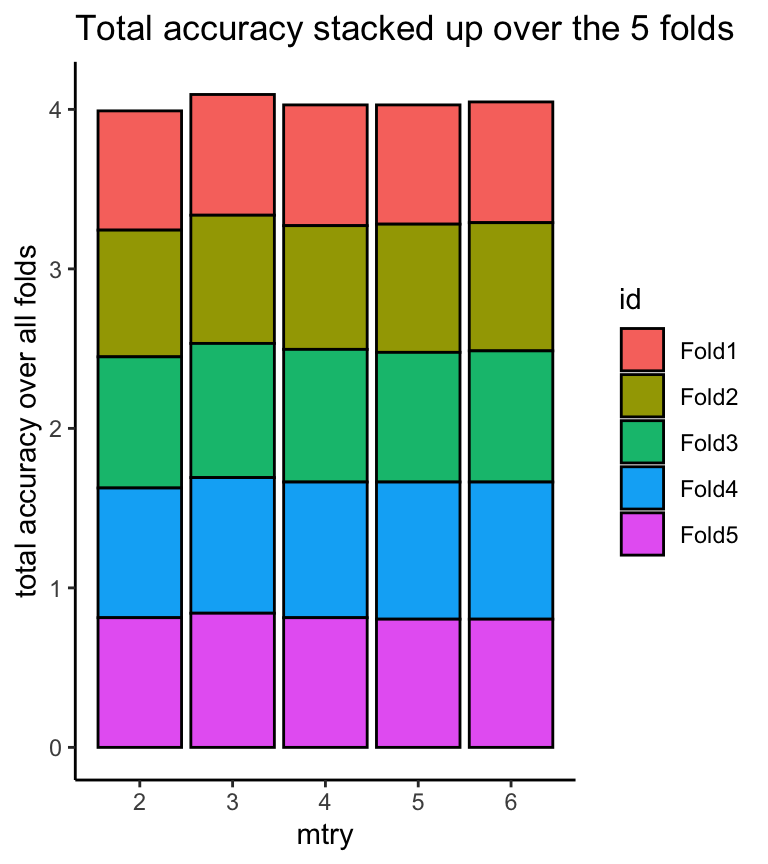
12.4.6 Lave final model med titanic datasæt
I min final model anvender jeg parametrene jeg valgt i min cross-validation proces, og træner jeg min model over hele titanic_training datasæt på én gang.
final_RF <- ranger(formula = Survived ~ . , data = titanic_training,
mtry = 3,
num.trees = 100)Nu jeg kan endelige anvende modellen til at lave forudsigelser over min titanic_test datasæt. Så kan jeg får en “final” accuracy, som fortæller hvor god min model er at finde ud af hvem der overlevede tradgedien, baserede på de andre variabler.
predictions <- tibble("actual"=titanic_test$Survived,
"predicted"=predict(final_RF,titanic_test)$predictions)
predictions %>% summarise("accuracy"=sum(actual==predicted)/n())## # A tibble: 1 × 1
## accuracy
## <dbl>
## 1 0.821Så kan man se, at vi er faktisk god til at forudsige hvem, der overlevde tradgedien (se også de forskellige scores og analyser på Kaggle).
12.4.7 Variable importance
Den allersidste, som kan være nyttig at vide, er hvilke variabler var mest vigtig for at kunne lave en nøjagtig forudsigelse af hvem, der overlevede. Man kan tilføje en indstilling, der hedder importance = "impurity" til kommandoen i funktion ranger, som i følgende:
final_RF <- ranger(formula = Survived ~ . , data = titanic_training,
importance = "impurity",
mtry = 3,
num.trees = 100)Sidste vil vil gerne vide, bidragen af de forskellige variabler i modellen:
mod <- t(final_RF$variable.importance) %>% as_tibble()
mod <- mod %>%
pivot_longer(everything())
mod## # A tibble: 6 × 2
## name value
## <chr> <dbl>
## 1 Pclass 29.7
## 2 Sex 59.0
## 3 Age 62.6
## 4 Solo 6.10
## 5 Port 5.78
## 6 Fare 68.1Den højere værdien, jo vigtigere er variablen:
mod %>%
ggplot(aes(x=name,y=value,fill=name)) +
geom_bar(stat="identity",colour="black") +
scale_x_discrete(limits=mod %>% arrange(desc(value)) %>% pull(name)) +
theme_bw()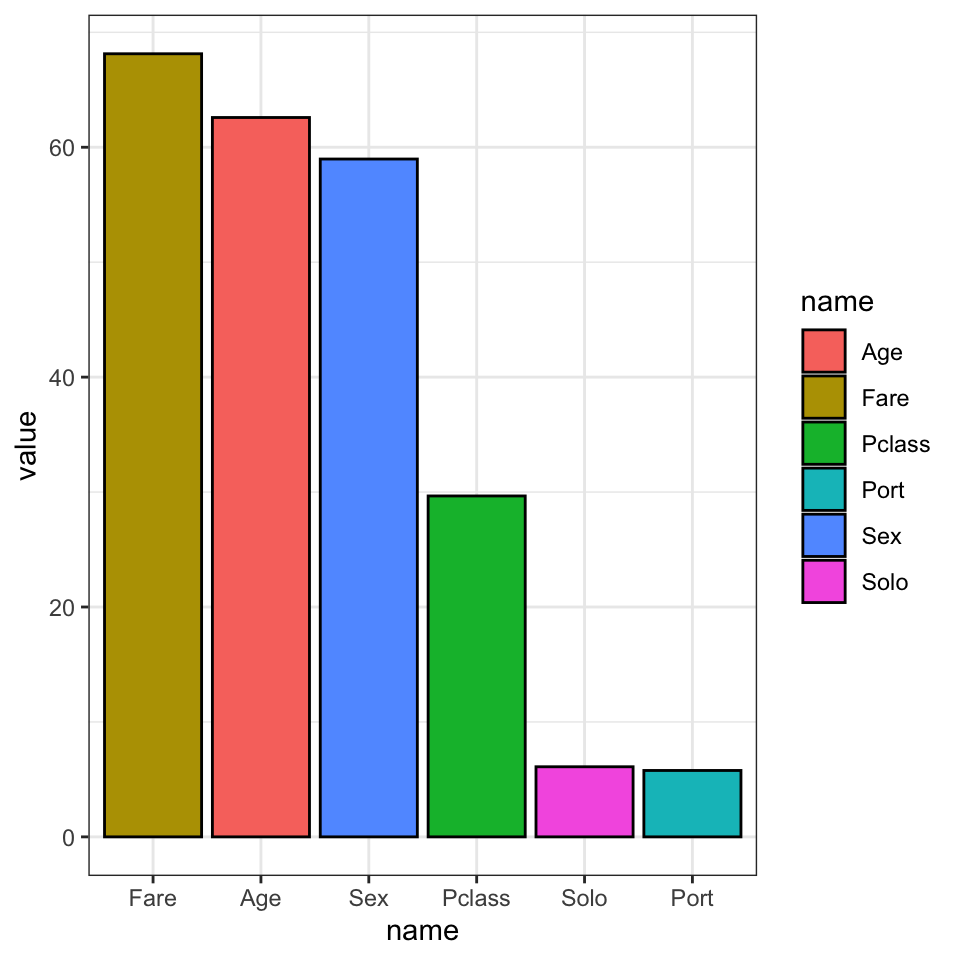
Så kan man se, at variablen Sex er meget vigtig for at udgøre hvem, der overlede, hvilket er i ovenenstemmelse med vors undersøgelser med titanic fra tidligere emner!
12.5 Problemstillinger
Problem 1): Quiz - maskinslæring
Classification med penguins
I datasættet “penguins” vil vi gerne anvende en classification model for at forudsige “male” og “female” pengvinger (variablen sex) ud fra de andre variabler i datasættet. Første åbne datasættet:
library(palmerpenguins)
penguins <- penguins %>% drop_na()OBS: Vi tilpasser koden tæt fra ovenstående eksempel med datasættet titanic - i følgende spørgsmål følg med koden fra Section 12.4
Lad os begynde med at opdele datasættet i to og brug testing data til at lave en “nested” dataframe som vi kan bruge til at lave 5-fold-cross-validation.
Problem 2) Set up cross-validation framework
a) Opdel datasættet “penguins” i to, således at 80 procent af observationerne hører til det træning datasæt (og 20 procent hører til det testing datasæt). Kald dit resultat penguins_split. Hvor mange observationer er blevet allokeret til træning og testing datasæt?
b) Udtræk de to datasæt fra penguins_split og kalder dem for penguins_training og penguins_test.
Vi lægger datasættet penguins_test til siden (vi får bruge for det senere), og arbejder kun med datasættet penguins_training for at udvikle vores model.
c) Anvend penguins_training til at lave en 5-fold-cross-validation dataframe, der hedder cv_split.
d) Udtræk de fem testing og training datasæt fra kolonnen splits og lagr dem i egne kolonner (kald deres kolonner for henholdvis validate og train). Kald din nye “nested” dataframe for cv_data.
Nu vil vi gerne lave samme model 5 gange - en gang til hvert datasæt i kolonnen train i vores nye cv_data dataframe. Den model skal være en ‘classification’ model, når vi gerne vil forudsige “male” eller “female” pingviner (vores afhængig variabel er sex som er kategorisk med to mulige værdier).
Problem 3) Set-up modellen
a) Lad os starte med det første datasæt og generaliser bagefter til en custom funktion i b). Anvend pluck for at få frem til det første datasæt i kolonnen train, og dernæst anvende funktionen ranger for at lave en classifikation model med variablen sex som afhængig variable, og med alle de andre variabler som uafhængige variabler (betegnet med ~ . i modellen). Angiv num.trees=100 (vi ignorerer andre mulige parametre indtil videre).
b) Når vi har fået modellen med ranger til at virke på det første datasæt laver vi en custom funktion der kan anvendes over samtlige datasæt i kolonnen train - tilpas din ranger model fra a) til en custom funktion og kalder den for ranger_function.
c) Anvend ranger_function på samtlige datasæt i kolonne “train” (opret en ny kolonne icv_data, der hedder model hvor dine fem modeller bliver lagret).
Nu har vi lavet en classification model til hvert træning sæt! Vi vil gerne vide, hvor gode modellerne er til at forudsige sex i de fem datasæts i kolonnen validate (husk at validate indeholder det styk, der ikke var brugt i training til den relevant id(fold)). Vi skal sammenligne to ting fra vores validate sæt - hvad er vores forudsigelser ud fra modellerne (“male” eller “female” til hver pingvin), og hvad er de virkelige værdier for variablen sex (igen “male” eller “female”).
Problem 4) Make predictions from model
a) Brug map/map2 til at lave to nye kolonner i cv_data:
actualhvor du udtrækker de virkelige værdier for variablensexpredictionhvor du anvenderpredictmed din models (betegnet som .x) og dine validate sæt (betegnet som .y)
b) Udvælg id, actual og prediction, unnest og beregn “accuracy” (proportionen svar, der er rigtige) til hvert af de 5 folds.
c) Lav et barplot for at se din accuracies over de 5-folds.
Nu har vi lavet 5-fold-cross validation til at træne en classification model. Vi kunne godt tænke os at udvide processen lidt, fordi der er nogle parametre der kan ændres på i funktionen ranger. Det er gennem den her ‘udviklingsproces’ hvor vi begynder at forstå fordelen af at bruge cross-fold-validation. Lad os prøve at ændre på parameten mtry for at se, om vi kan gøre modellen bedre. Igen følger vi meget tæt på ovenstående kurusnotaterne.
Problm 5) Tune parameteren mtry.
a) Anvend funktionen crossing til at inddrage mtry (fra 2 til 6 som er antal uafhængig variabler) som kolonne i cv_data. Kald din nye dataframe for cv_tune. Hvor mange datasæt er der nu i kolonnen train?
b) Tilpas din custom ranger funktion fra Spørgsmål 3 hvor du nu inddrager parameteren mtry (med værdi betegnet som .y) og anvende din model på de forskellige training sæt i kolonnen train for hver mulig værdi af mtry (husk at bruge map2 her).
c) Lav forudsigelser ud fra samtlige modeller og dine validation sæt (kolonnen validate) og udtræk både dine “actual” og “predicted” værdier præcis ligesom før.
d) Beregn accuracy for samtlige sæt (alle kombinationer af id og mtry), lav et stacked barplot og vælg den bedste mulige værdi for mtry.
Nu at vi har valgt en værdi for parameteren mtry, lad os går henne og lav en “final” model! Når vi har lavet den final model kan vi endelig kigge på vores penguins_test datasæt, som vi lagt tidligere til siden. Husk at validate sæts var brugt til at teste modellen undervejs i udviklingsprocessen, men de var stadig en del af penguins_training. Man kigger på den testing datasæt kun én gang til sidste for at spørge - hvor god er vores endelige model?
Problem 6) Lave en final model
a) Anvende funktionen ranger til at lave en classifcation model på datasættet penguins_training. Angiv num.trees=100 og din valgt værdi for parameteren mtry.
b) Lav predictions med funktionen predict hvor du angiv din “final” model samt dit “penguins_test” datasæt. Beregen accuracy fra din model ved at sammenligne de virkelige værdier for variablen sex i dit testing sæt med din beregnede værdier fra predict().
Nu at vi er færdig med at lave og bedømme vores “final” model er vi interesseret i - hvilke variabler havde den største indflydelse på vores forudsigelser af “male” og “female” pingviner?
Problem 7) Variable importance
a) Tag din funktion til din “final” model og angiv indstillingen importance = "impurity" i funktionen. Kør modellen og udtræk din variable importance vector med final_RF$variable.importance.
b) Omsæt din importance scores til et barplot hvor du angiv rækkefølgen af søjlerne efter vigtigheden af de forskellige variabler (den meste vigtig til venstre).
Problem 8) Gentag samme process i et andet datasæt men i en regression contekst:
LungCapData <- read.csv("https://www.dropbox.com/s/ke27fs5d37ks1hm/LungCapData.csv?dl=1")
glimpse(LungCapData)## Rows: 725
## Columns: 6
## $ LungCap <dbl> 6.475, 10.125, 9.550, 11.125, 4.800, 6.225, 4.950, 7.325, 8.…
## $ Age <int> 6, 18, 16, 14, 5, 11, 8, 11, 15, 11, 19, 17, 12, 10, 10, 13,…
## $ Height <dbl> 62.1, 74.7, 69.7, 71.0, 56.9, 58.7, 63.3, 70.4, 70.5, 59.2, …
## $ Smoke <chr> "no", "yes", "no", "no", "no", "no", "no", "no", "no", "no",…
## $ Gender <chr> "male", "female", "female", "male", "male", "female", "male"…
## $ Caesarean <chr> "no", "no", "yes", "no", "no", "no", "yes", "no", "no", "no"…Følg processen fra Sektion 12.3 igennem med datasættet LungCapData:
- forudsige
LungCapsom afhængig variabel og alle andre variabler som uafhængig variabler - Splitte datasættet (75% training, 25% testing) og anvend 10-fold cross-validation på dit training datasæt
- Beregn “mean absolute error” for dine forskellige folds
- Lav en færdig model og beregn “mean absolute error”
12.6 Yderligere kommentarer og pakker
Jeg spang over mange nyttige pakker og funktioner, der kan bruges til at lave maskinslæring processer i R
Yardstick-pakken er til metrics - fk. accuracy/precesion/recall osv. man kan også lave ROC curves osv. Jeg anvendte kun accuracy i kurset (til at vise processen/iden men begrænser overordnet læs i emnet) men man plejer at anvende forskellige metrics i virkeligheden.Tidymodels-pakken er en generel pakke, hvor man få en “fælles” tilgang til at opbygge modeller der stammer fra mange forskellige pakke - man kan fk. bruge samme tilgang til at lave blandt andet regression eller bayesian modeller, random forest, support vector machines osv. - alle modeller har nu samme syntaksen i denTidymodelstilgang.- Der er mange bøger der handler om machine learning n the tidyverse, fk. se her https://livebook.manning.com/book/machine-learning-for-mortals-mere-and-otherwise/chapter-1/v-4/1